材料与方法:
1.中药制剂成分组成
药方:**基 于 客 户 提 供 的 中 药 名 称 , 使 用 TCMSP 数 据 库(http://lsp.nwu.edu.cn/browse.php?qc=herbs)[1]获得药物成分信息,包括此味药包含多少种成分、Molecule Name、MW(molecular weight)、AlogP(Lipid/ waterPartition Coeffici)、Hdon/Hacc(hydrogen-bond donors / acceptors)、OB(Oralbioavailability)、Caco-2(intestinal epithelial permeability)、BBB(blood-brainbarrier)、DL(Drug-likeness)、HL(Drug half-life)等。
2.药物有效成分筛选
使用 ADME[2,3]参数筛选出可能的药物小分子,“ADME”即“毒药物动力学”,指机体对外源化学物的吸收(absorption)、分布(distribution)、代谢(metabolism)及排泄(excretion)过程。外源化学物的代谢和排泄合成为消除,吸收、分布、代谢和排泄的过程可能同时发生。ADME 数据来源于 TCMSP 数据库。ADME 参数包括口服生物利用度(OB),药物相似性(DL)以及药物半衰期(HL)等。这些参数均为预测值,TCMSP 推荐筛选阈值(可调整):OB: ≥30%;DL ≥0.18;BBB: <-0.3 is non-penetrating (BBB-), from -0.3 to +0.3, moderate penetrating(BBB±),and>0.3 strong penetrating (BBB+);HL: Drug half-life ≤4 hours: fast-elimination group, between 4-8 hours are mid-elimination group and ≥8 hours are slow-elimination group;主要常规参数进行药物成分筛选,阈值为 OB≥40%;DL≥0.2。
3.成分靶蛋白预测筛选
基于 TCMSP 和 DRUGBANK 数据库[4](https://www.drugbank.ca/),预测化学小分子对应的靶蛋白,DrugBank 数据库提供的是已经实验验证、临床或者已经上市药物的相关信息,结果准确可靠。靶蛋白名称为全称,通过 uniprot 数据库(https://www.uniprot.org/)的 uniprotID 对应至基因简称 gene symbol。
4. 靶蛋白交叉验证
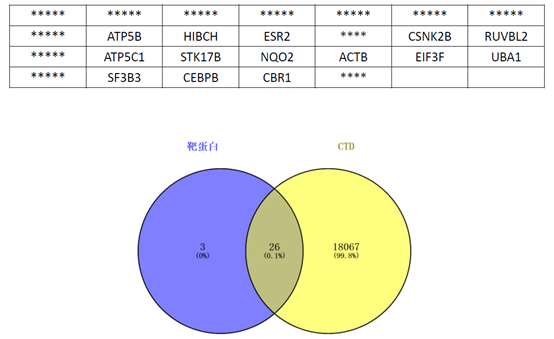
Mount Desert Island Biological Laboratory 已经公开了 CTD 2018 update(Comparative Toxicogenomics ,http://ctdbase.org/)[5],旨在促进对环境风险如何影响人类健康的了解,它提供了关于化学-基因/蛋白质相互作用、化学-疾病和基因-疾病关系的信息,以帮助制定关于环境影响的疾病机制。在 CTD 数据库中以“*********”为关键词搜索****相关基因,并与上一步所预测的靶蛋白取交集以缩小靶蛋白范围。
5.成分靶蛋白 GO 功能分析、通路分析
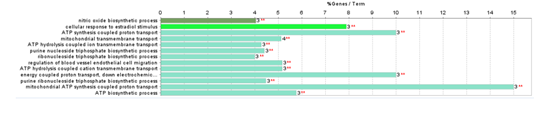
基于 cytoscape 插件 ClueGO + CluePedia [6]对靶蛋白进行 GO 功能[7]分析,设置选 GO biological Process,显著性阈值设为 P.adjust≤0.05。并用 cytoscape 做靶蛋白功能网络图。气泡图进行可视化。
6.成分靶蛋白互作分析
我们利用 STRING(version:10.0, http://www.string-db.org/)数据库[9]对mRNA 进行蛋白质与蛋白质相互作用关系分析(Protein-Protein Interaction,PPI),选择 Required Confidence (combined score) > 0.4 为蛋白质与蛋白质相互作用关系的阈值,并下载相关 tsv 格式文件。得到 PPI 关系对文件后,使用 Cytoscape 软件对其进行网络图构建。
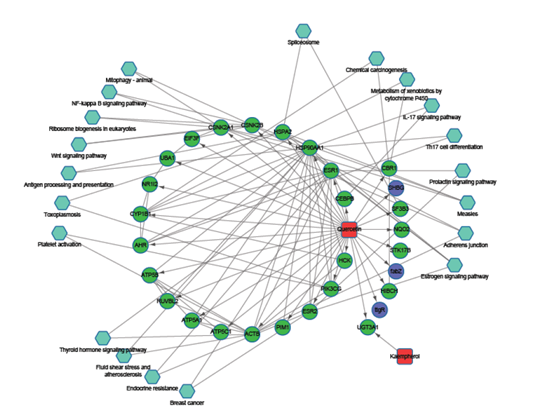
7. 网络药理学网络构建
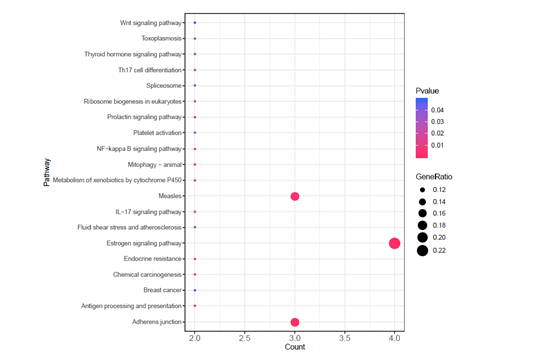
基于成分-靶蛋白-通路利用 cytoscape 构建药理学网络。展示**中重要成分
的****相关靶蛋白的通路调控机制。
8. 靶蛋白 GEO 数据集验证
取一套 GEO 数据库****mRNA 芯片数据剔除23****样本。下载****** 数据集原始数据文件,利用R 包affy 包(Version 1.50.0,http://www.bioconductor.org/packages/release/bioc/html/affy.html ) 进 行读 取 , 以RMA(robust multi-array average)方法进行标准化预处理,包括背景校正、标准化、归一化。利用平台注释文件对探针进行注释,去除没有匹配到基因(Genesymbol)的探针;对于不同探针映射到同一基因,我们取不同探针的均值作为这个基因最终的表达值。
将样本分为两组,疾病组和健康组,对 OA(疾病)vs Ctrl(健康)样本的表达,利 用 limma 包 [10] ( Version 3.26.9 )计算表达差异性 p值和表达 Fold Change 值。
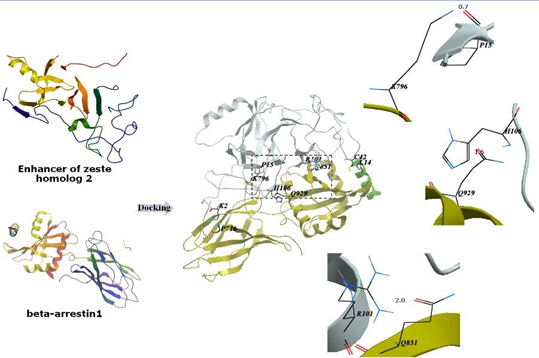
9. 关键靶蛋白-成分分子对接预测
与靶蛋白取交集,通过 PDB 数据库[11]搜索关键靶蛋白的 PDB ID,PDB 数据库提供关于蛋白质、核酸和复杂组件的 3D 形状的信息,帮助研究人员理解生物 医 学 和 农 业 方 面 的 生 物 学 信 息 。 我 们 通 过 pymol(Version 2.1.1,https://pymol.org/dokuwiki/?id=) 软 件 对 关 键 蛋 白 进 行 3D 结 构 展 示 , 通 过SystemsDock 在线工具[12,13]预测与**重要成分的对接模型得分 pKd/pKi(实验
离解/抑制常数值),即预测的结合亲和力,得分区间(1~10)得分越高表示,
绘制蛋白质-配体相互作用示意图。**重要成分化学式从 Drugbank 数据库下载。
1.中药制剂成分组成及成分筛选
表 1 **中重要化学成分
Mol ID Molecule Name OB DL
********** ************* 42.98 0.76
********** ************* 41.75 0.4
********** ********** 44.23 0.82
********** ********** 40.12 0.75
********** ********** 43.09 0.78
MOL000422 *kaempherol 41.88 0.24
2.成分靶蛋白预测筛选
3. ****靶蛋白交叉验证
4. ****靶蛋白GO功能分析、通路分析
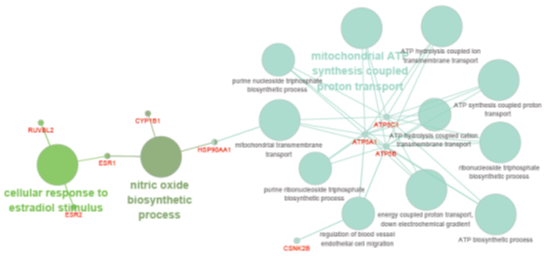
5.靶蛋白相互作用分析
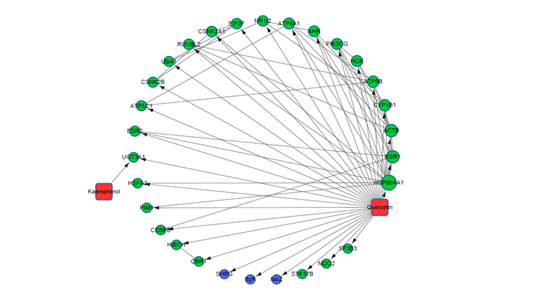
6. 网络药理学网络构建
7. 分子对接
参考文献
[1] Jinlong Ru; Peng Li; Jinan Wang; Wei Zhou; Bohui Li; Chao Huang; Pidong Li; Zihu Guo;Weiyang Tao; Yinfeng Yang; Xue Xu; Yan Li; Yonghua Wang; Ling Yang. TCMSP: a of systems pharmacology for drug discovery from herbal medicines. JCheminformatics 2014 Apr 16;6(1):13.
[2] Madden, J. C. (2010) In silico approaches for predicting ADME properties. Recent advances in QSAR studies .Springer, pp. 283 304.
[3] Ertl, et al. (2000) Fast calculation of molecular polar surface area as a sumof fragment b ased contributions and its application to the prediction of drug transport properties. J. Med. Chem. 43 , 3714 3717.
[4]Wishart DS, Feunang YD, Guo AC, Lo EJ, Marcu A, Grant JR, Sajed T, Johnson D, Li C,Sayeeda Z, Assempour N, Iynkkaran I, Liu Y, Maciejewski A, Gale N, Wilson A, Chin L,Cummings R, Le D, Pon A, Knox C, Wilson M. DrugBank 5.0: a major update to the DrugBank for 2018. Nucleic Acids Res. 2017 Nov 8. doi: 10.1093/nar/gkx1037.
[5] Davis AP, Grondin CJ, Johnson RJ, Sciaky D, McMDavis AP, Grondin CJ, Johnson RJ, Sciaky D, McMorran R, Wiegers J, Wiegers TC, orran R, Wiegers J, Wiegers TC, Mattingly CJ The Comparative Toxicogenomics : update 2019. Nucleic Acids Res. Mattingly CJ The Comparative Toxicogenomics : update 2019. Nucleic Acids Res. 2018 Sep 24.2018 Sep 24.
[6] Bindea G*, Mlecnik B*, Hackl H, Charoentong P, Tosolini M, Kirilovsky A, Fridman Bindea G*, Mlecnik B*, Hackl H, Charoentong P, Tosolini M, Kirilovsky A, Fridman WH
Pages F, Trajanoski Z and Galon J. ClueGO: a Cytoscape pluglueGO: a Cytoscape plug--in to decipher functionallyin to decipher functionally
grouped gene ontology annotation networks. Bioinformatics 2009.25(8):1091
7 ] M Ashburner, C A Ball, J A Blake, D Botstein, H Butler, J M Cherry, A P Davis, KDolinski, S S Dwight, J T Eppig, M A Har ris, D P Hill, L Issel Tarver, A Kasarskis, S Lewis, JC Matese, J E Richardson, M Ringwald, G M Rubin, G Sherlock. Gene ontolo gy: tool for the
unification of biology The Gene Ontology Consortium. Nat Genet, 25:25 29, 2000.
[8 ] da W Huang, B T Sherman, Q T an, J R Collins, W G Alvord, J Ro ayaei, R Stephens, M WBaseler, H C Lane, R A Lempicki. The DAVID Gene Functional Classifi cation Tool: a novel biological module centric algorithm to functionally analyze large gene l ists. Genome Biol,
8:R183 R183, 2007.
[9]Szklarczyk et al. Nucleic Acids Res. 2015 43( issue):D447 5210 Smyth, G.K., Limma: linear models for microarray data, in Bioinformatics and computational biology solutions using R and Bioconductor. Springer, 2005: p. 397 420.
[10]Smyth, G.K., Limma: linear models for microarray data, in Bioinformatics andcomputational biology solutions using R and Bioconductor. Springer, 2005: p. 397 420.
[11]H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig , I.N.Shindyalov, P.E. Bourne. Bourne.(2000) The Protein Data Bank Nucleic Acids Research, 28: 235 242.
[12]Hsin, K. Y., Matsuoka, Y., Asai, Y., Kamiyoshi, K., Watanabe, T., Kawaoka, Y. andKitano, H. (20 16) systemsDock: a web server for network pharmacology based prediction and analysis. Nucleic Acids Research (DOI: 10.1093/nar/gkw335)
[13]Hsin K. Y., Ghosh, S., Kitano, H. (2013) Combining machine learning systems and multiple docking simulation packages to improve docking prediction reliability for network pharmacology. PLOS ONE(DOI: 10.1371/journal.pone.0083922)